Las moléculas de ARN mensajero utilizadas en diferentes terapias (vacunas contra agentes infecciosos, tratamientos para el cáncer) están formadas por una caperuza (CAP) en el extremo 5′ que minimiza su degradación y facilita el proceso de traducción al unirse a los ribosomas de la célula.
De igual modo el extremo 3′ también está protegido, en este caso por una cola de adeninas (Poli-A), que consta de entre 100 y 150 nucleótidos de adenina y evita la degradación de la molécula por parte de las ribonucleasas (RNasa).
Tanto la caperuza como la cola poli-A se añaden al ARN por métodos enzimáticos, es decir, no están codificados en el molde de ADN a partir del cual se obtienen las copias de ARN. La poliadenilación enzimática genera colas poli-A de tamaños muy variables, lo cual tiene una gran influencia en la eficiencia de la traducción que producirá la proteína deseada. ARN con colas muy cortas se degrada rápidamente y no llega a traducirse en una proteína.
Por tanto, es necesario desarrollar técnicas analíticas que permitan conocer la longitud de la cola poli-A del ARN terapéutico. ¿Cómo se hace?. Estoy seguro de que algunos nos propondrían comprar un microscopio, pero las moléculas, incluso las grandes, están fuera del alcance de estos aparatos (aún siendo electrónicos).
El análisis de mezclas complejas, como las vacunas, precisa de métodos analíticos instrumentales que primero separen la mezcla en sus componentes y después puedan identificarlos y cuantificarlos. Una de las técnicas más poderosas de las que se dispone hoy en día es la cromatografía acoplada a espectrometría de masas. El cromatógrafo líquido (HPLC) separa los componentes, que van siendo suministrados al espectrómetro de masas el cual identifica y cuantifica cada uno de ellos.
Estas técnicas analíticas sólo están al alcance de universidades y laboratorios de algunas industrias ya que el conjunto de ambos equipos (LC-MS) supera los 100 000 dólares. El resto de los mundanos se tienen que conformar con el microscopio de 40 aumentos.
La determinación de la longitud de la cola poli-A se realiza rompiendo las moléculas de ARN con la ribonucleasa T1 que degrada la cadena por todas las posiciones que contengan el nucleótido de guanina. Dado que los ARN son muy ricos en guanina, la cadena rompe en muchísimos fragmentos de pocos nucleótidos cada uno, pero la cola poli-A no se fragmenta ya que no tiene guanina, está formada exclusivamente por adeninas.
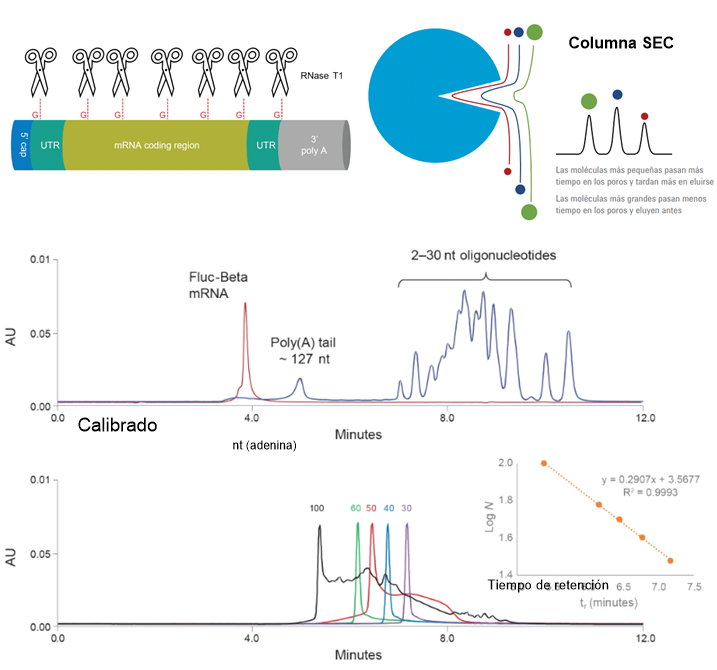
La columna cromatográfica separa los diferentes fragmentos según su tamaño, entre ellos están las colas poli-A que también serán separados según el número de nucleótidos que contengan. Los fragmentos más largos tienen tiempos de retención más cortos, al interaccionar menos con la fase estacionaria de la columna. Los fragmentos pequeños interaccionan más y el tiempo de retención es mayor, tardan más en salir de la columna. Mediante un calibrado se puede asociar el tiempo de retención al tamaño, lo que permite conocer el número de nucleótidos de adenina que corresponden a cada pico del cromatograma.
Los problemas analíticos no terminan con la cola poli-A, es necesario comprobar que la mayor parte de las moléculas poseen la caperuza, el grado de degradación del ARN (algunas moléculas se rompen), el tamaño de las nanopartículas lipídicas, así como posibles impurezas.
Como podéis ver la cosa es más compleja que buscar óxido de grafeno con microscopios ópticos.
https://www.nature.com/articles/s41565-022-01129-w
https://www.agilent.com/cs/library/primers/public/5991-3651ES.pdf
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8450916/

